Qui est en ligne ?
Il y a en tout 33 utilisateurs en ligne :: 0 Enregistré, 0 Invisible et 33 Invités :: 1 Moteur de rechercheAucun
Le record du nombre d'utilisateurs en ligne est de 480 le Jeu 16 Sep - 4:29
Derniers sujets
Taxonomie bacterienne
DOC DZ...La Communauté des Etudiants et Praticiens en Médecine :: FORUMS VOISINS ... :: FILIERES VOISINES
:: les biologistes :: Taxonomie
Page 1 sur 1
 Taxonomie bacterienne
Taxonomie bacterienne
Taxonomie
La taxonomie des bactéries soulève de grandes questions: tout d'abord, qu'est-ce qu'une espèce en bactériologie et comment la déterminer ? Ensuite, comment comparer et classer les bactéries ?
Taxonomie
La taxonomie a pour objectif de nommer, classer et identifier les organismes.
1. Nommer (nomenclature latine binomiale). Exemple: Escherichia coli K12: le nom de genre est indiqué en premier et doit posséder une majuscule. Le nom d'espèce est binomial, comprenant un second terme en minuscule. La souche, sous-unité de l'espèce, est indiquée ensuite (ici souche K12).
2. Classer. Une espèce n'a pas de nom tant qu'elle n'a pas pu être isolée et caractérisée. Initialement, il fallait la cultiver en culture pure. Mais celà ne concernait qu'une infime partie des micro-organismes présents sur Terre ! Avec les progrès de la biologie moléculaire, il est désormais possible d'isoler des séquences d'organismes inconnus. Comme nous le verrons plus loin, il est possible de classer par phylogénétique ou par phénotypie, afin de réaliser une classification de différentes bactéries en cultures pures.
3. Identifier une espèce, au sens de découvrir cette espèce, se fait par des indices phénotypiques ou génétiques.
La notion d'espèce
La notion d'espèce est définie en biologie animale par le critère d'interfécondité: deux individus sont de la même espèce si leur descendance est fertile.
Cette définition s'applique très souvent aux Vertébrés, car elle se base sur des barrières immunologiques.
Pour les plantes, les horticulteurs connaissent fort bien les hybrides; et pour les micro-organismes, comment inclure le résultat d'une reproduction sexuée lorsque la reproduction est très majoritairement clonale, comme dans le cas des bactéries ?
En taxonomie, cette question pose un problème de raisonnement, et il faut établir d'autres méthodes de comparaison, plus fiables et plus universelles. Reprenons le problème: nous avons 2 bactéries, A et B, en cultures pures. On souhaite étudier leur taxonomie. Nous avons alors trois cas posibles:
* A = B et les deux bactéries sont de la même espèce
* A proche de B et les espèces sont voisines (E. coli et Salmonella typhimurium)
* A différent de B (E. coli et Mycoplasma sp.)
Pour comparer A et B, il va falloir rechercher leurs similitudes et leurs différences. pour se faire, on peut procéder par comparaison de caractères ou par recherche de relations évolutives.
Il est possible d'établir une classification phénotypique ou phénétique en comparant les caractères phénotypiques lourds (types trophiques, parois, ...) et légers (enzyme isolée) entre deux bactéries A et B.
La recherche de liens évolutifs entre espèces aboutit à une classification phylogénétique. Cette méthode utilisait autrefois pour seuls indices les fossiles. Mais ceci n'est pas applicable aux bactéries comme en paléontologie des Vertébrés par exemple ... La classification phylogénétique a été facilitée grâce aux progrès de la biologie moléculaire, qui fait des acides nucléiques des "indice évolutif" désormais largement utilisés !
Les acides nucléiques peuvent être donc utilisés aussi bien en classification phénotypique qu'en classification phylogénétique :
Pour étudier les similitudes d'ordre génétiques, on utilise les méthodes suivantes:
*Physiques: la température de demi-dénaturation (température de fusion) de l'ADN donne une indication sur le % de G+C: plus il est haut, plus le % est fort. Par ultracentrifugation isopycnique sur gradiant CsCl, on peut avoir une comparaison par densité de flottaison.
* Chimiques: dosages au spectrophotomètre donne des valeurs sur le % de G+C. L'hybridation entre l'ADN de la bactérie A et l'ADN de la bactérie B pour donner des réapparillements hybrides, cette méthode utilisée via une chromatographie, en fixant un des deux ADN et en marquant radioactivement l'ADN libre permet de savoir s'il y a hybridation ou non. On parle alors de RBR (% de réappariement). Enfin par des comparaisons de séquence (via séquençage), on peut connaître le % de similitude séquentielle S.
Classification phénotypique
Cette classification se base, comme vu précédemment, sur des critères morphologiques, structuraux, fonctionnels des bactéries.
Les taxons permettent de regrouper l'ensemble des organismes ayant le même ancêtre commun: ces paliers dans la classification permettent de remonter l'arborescence du Vivant de la manière suivante:
* Règne: Procaryotes
* Embranchement / Division: Gracilicutes
* Classe: Scotobacteriae
* Ordre: Enterobacteriales
* Famille: Enterobacteriaceae
* Genre: Escherichia
* Escherichia coli
NB: Ces données correspondent à la classification phénotypique !
Comment sont classées les bactéries dans cette classification ? En fait, il faut remarquer que les 4 divisions sont bâties selon des différences sur la structure de l'enveloppe, la physiologie et le mode de vie:
1. Gracilicutes (eubactéries gram -, deux membranes)
* Anoxyphotobacteriae
(photosynthèse sans production d'oxygène)
* Photobacteriae
(cyanobactéries) * Scotobacteriae
(pas de photosynthèse)
2. Firmicutes (eubactéries gram +, une membrane)
* Firmibacteriae
(% G+C faible)
* Thallobacteriae
(%G+C fort, ramifiées)
3. Tenericutes
* Mollicutes
(eubactéries sans paroi, gram -)
4. Mendosicutes
* Archaebacteriae
(archéobactérie gram + / -)
Quels outils pour comparer les bactéries entre elles ? Une méthode largement employée autrefois, mais désormais critiquée et soumise à réserves, est la taxonomie numérique.
Cette approche quantitative de la taxonomie est définie par Peter H. A. Sneath et Robert Sokal comme "le groupement d'unités taxonomiques, en taxons à l'aide de méthodes numériques sur la base des états de leurs caractères".
La méthode consiste à réaliser un tableau de caractères, et à indiquer si chaque bactérie le possède ou non. Puis, à partir de ces données, on réalise une comparaison et des calculs de similarité. Prenons un exemple de calcul: le pourcentage de simple appariement:
Calcul des coefficients d'association de deux organismes:
Soit 4 ensembles a, b, c, d et deux bactéries isolées A et B:
* a: caractères présents chez A et B. (en rouge)
* b: caractères propres à la bactérie A. (bleu)
* c: caractères propres à la bactérie B. (vert)
* d: caractères manquant aux deux bactéries (beige)

Notion d'association de caractères
Simple appariement :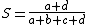
% de similarité :
Coefficient de Jaccard:
Les résultats sont exprimés sous forme de dendrogrammes: classification ascendante hiérarchique par niveau d'intégration.
Classification phylogénétique
En 1965, Pauling et Zuckerhandl suggèrent que les séquences d'acides nucléiques et protéiques changent avec le temps, et peuvent être considérées comme des chronomètres moléculaires.
Grâce à ce concept, il est donc possible de réaliser un arbre phylogénétique à partir de la comparaison de séquences définies chez différents organismes. En effet, on suppose qu'au cours du temps, ces séquences on été graduellement modifiées, si bien que des fonctions ont pu être perdue ou modifiées. Des séquences similaires se révélant très différentes chez deux groupes comparés montrent donc que ces deux groupes ont divergé voilà longtemps.
on choisira des séquences conservées chez tous les organismes pour réaliser une classification des grands groupes, et des séquences plus spécifiques pour réaliser une classification plus précise sur une famille, un genre, des populations ...
Pour les bactéries, nous savons que leurs ribosomes se composent de différents ARN ribosomiques, dont l'ARN 16S (S = Svedberg). Ni trop petit ni trop long, il a été choisi comme élément de comparaison au tout début de la taxonomie moléculaire. Il est digéré par la RNAse T1, et les fragments obtenus furent analysés en premier par Woese, obtenant l'arbre de Woese (voir figures ci-dessous):
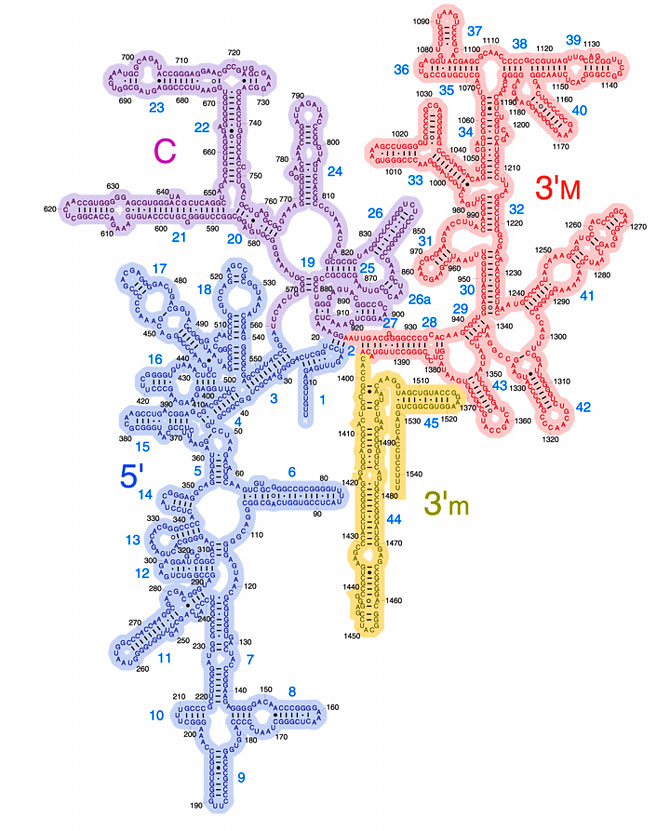
ARN ribosomal 16S
A partir de ces comparaisons, il a été possible de dessiner un premier arbre phylogénique moléculaire. Woese réalisa cette construction:
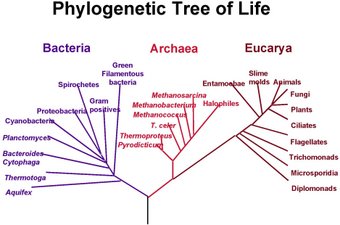
Arbre phylogénétique universel
Etabli par comparaison de séquences d'ARNr
La position exacte des trois ensembles sur l'arbre est encore discutée, comme nous aurons occasion de le voir ultérieurement dans un article consacré au sujet, mais en attendant le lecteur intéressé par cet arbre peut se rapporter à l'article de G. J. Olsen & C.R. Woese: Ribosomal RNA: a key to Phylogeny. The FASEB Journal, 7, pp. 113-123, 1993.
Dans un arbre phylogénétique, on appelle noeud chaque point correspondant à un organisme ancêtre et branches les cheminements évolutifs ayant mené aux noeuds-fils. L'arbre peut avoir une échelle de temps ou de modifications moléculaires, alors représentées par la longueur des branches. Il peut être enraciné (montre un ancêtre commun) ou non (mettant en évidence uniquement les relations phylogénétiques).
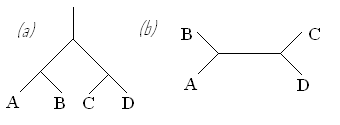
Exemples d'arbres phylogénétiques enracinés (a) ou non (b)
Les résultats sont différents de la classification phénotypique ! Voici de nouveau l'exemple d'Escherichia coli:
* Règne: Procaryotes
* Embranchement / Division: Proteobacteria
* Classe: Gammaproteobacteriae
* Ordre: Enterobacteriales
* Famille: Enterobacteriaceae
* Genre: Escherichia
* Escherichia coli
Ces données ont été obtenues à partir de Pubmed Taxonomy browser.
Nous avons vu plus haut des exemples de protocoles expérimentaux et d'interprétation des résultats concernant la similitude entre deux bactéries A et B. Les caractères génétiques ainsi isolés peuvent également être retenus dans des matrices et donner des dendrogrammes !
La taxonomie polyphasique
De nombreux taxonomistes pensent que seule une combinaison des données génotypiques et phénotypiques peut permettre d'établir une phylogénie. C'est l'approche polyphasique.
Quand celà est possible, les taxonomistes s'emploient à utiliser un maximum d'informations, que ce soit des caractères écologiques, morphologiques, métaboliques comme des propriétés moléculaires, afin d'obtenir les résultats les plus fiables et les plus réalistes.
Sources:
* Prescott, Harley & Klein. Microbiologie. Editions DeBoeck (2003)
* Cours de microbiologie. Licence de Biochimie de Rennes 1 (2004)
* Pubmed taxonomy Browser (2006) [ En ligne ]
La taxonomie des bactéries soulève de grandes questions: tout d'abord, qu'est-ce qu'une espèce en bactériologie et comment la déterminer ? Ensuite, comment comparer et classer les bactéries ?
Taxonomie
La taxonomie a pour objectif de nommer, classer et identifier les organismes.
1. Nommer (nomenclature latine binomiale). Exemple: Escherichia coli K12: le nom de genre est indiqué en premier et doit posséder une majuscule. Le nom d'espèce est binomial, comprenant un second terme en minuscule. La souche, sous-unité de l'espèce, est indiquée ensuite (ici souche K12).
2. Classer. Une espèce n'a pas de nom tant qu'elle n'a pas pu être isolée et caractérisée. Initialement, il fallait la cultiver en culture pure. Mais celà ne concernait qu'une infime partie des micro-organismes présents sur Terre ! Avec les progrès de la biologie moléculaire, il est désormais possible d'isoler des séquences d'organismes inconnus. Comme nous le verrons plus loin, il est possible de classer par phylogénétique ou par phénotypie, afin de réaliser une classification de différentes bactéries en cultures pures.
3. Identifier une espèce, au sens de découvrir cette espèce, se fait par des indices phénotypiques ou génétiques.
La notion d'espèce
La notion d'espèce est définie en biologie animale par le critère d'interfécondité: deux individus sont de la même espèce si leur descendance est fertile.
Cette définition s'applique très souvent aux Vertébrés, car elle se base sur des barrières immunologiques.
Pour les plantes, les horticulteurs connaissent fort bien les hybrides; et pour les micro-organismes, comment inclure le résultat d'une reproduction sexuée lorsque la reproduction est très majoritairement clonale, comme dans le cas des bactéries ?
En taxonomie, cette question pose un problème de raisonnement, et il faut établir d'autres méthodes de comparaison, plus fiables et plus universelles. Reprenons le problème: nous avons 2 bactéries, A et B, en cultures pures. On souhaite étudier leur taxonomie. Nous avons alors trois cas posibles:
* A = B et les deux bactéries sont de la même espèce
* A proche de B et les espèces sont voisines (E. coli et Salmonella typhimurium)
* A différent de B (E. coli et Mycoplasma sp.)
Pour comparer A et B, il va falloir rechercher leurs similitudes et leurs différences. pour se faire, on peut procéder par comparaison de caractères ou par recherche de relations évolutives.
Il est possible d'établir une classification phénotypique ou phénétique en comparant les caractères phénotypiques lourds (types trophiques, parois, ...) et légers (enzyme isolée) entre deux bactéries A et B.
La recherche de liens évolutifs entre espèces aboutit à une classification phylogénétique. Cette méthode utilisait autrefois pour seuls indices les fossiles. Mais ceci n'est pas applicable aux bactéries comme en paléontologie des Vertébrés par exemple ... La classification phylogénétique a été facilitée grâce aux progrès de la biologie moléculaire, qui fait des acides nucléiques des "indice évolutif" désormais largement utilisés !
Les acides nucléiques peuvent être donc utilisés aussi bien en classification phénotypique qu'en classification phylogénétique :
Pour étudier les similitudes d'ordre génétiques, on utilise les méthodes suivantes:
*Physiques: la température de demi-dénaturation (température de fusion) de l'ADN donne une indication sur le % de G+C: plus il est haut, plus le % est fort. Par ultracentrifugation isopycnique sur gradiant CsCl, on peut avoir une comparaison par densité de flottaison.
* Chimiques: dosages au spectrophotomètre donne des valeurs sur le % de G+C. L'hybridation entre l'ADN de la bactérie A et l'ADN de la bactérie B pour donner des réapparillements hybrides, cette méthode utilisée via une chromatographie, en fixant un des deux ADN et en marquant radioactivement l'ADN libre permet de savoir s'il y a hybridation ou non. On parle alors de RBR (% de réappariement). Enfin par des comparaisons de séquence (via séquençage), on peut connaître le % de similitude séquentielle S.
Classification phénotypique
Cette classification se base, comme vu précédemment, sur des critères morphologiques, structuraux, fonctionnels des bactéries.
Les taxons permettent de regrouper l'ensemble des organismes ayant le même ancêtre commun: ces paliers dans la classification permettent de remonter l'arborescence du Vivant de la manière suivante:
* Règne: Procaryotes
* Embranchement / Division: Gracilicutes
* Classe: Scotobacteriae
* Ordre: Enterobacteriales
* Famille: Enterobacteriaceae
* Genre: Escherichia
* Escherichia coli
NB: Ces données correspondent à la classification phénotypique !
Comment sont classées les bactéries dans cette classification ? En fait, il faut remarquer que les 4 divisions sont bâties selon des différences sur la structure de l'enveloppe, la physiologie et le mode de vie:
1. Gracilicutes (eubactéries gram -, deux membranes)
* Anoxyphotobacteriae
(photosynthèse sans production d'oxygène)
* Photobacteriae
(cyanobactéries) * Scotobacteriae
(pas de photosynthèse)
2. Firmicutes (eubactéries gram +, une membrane)
* Firmibacteriae
(% G+C faible)
* Thallobacteriae
(%G+C fort, ramifiées)
3. Tenericutes
* Mollicutes
(eubactéries sans paroi, gram -)
4. Mendosicutes
* Archaebacteriae
(archéobactérie gram + / -)
Quels outils pour comparer les bactéries entre elles ? Une méthode largement employée autrefois, mais désormais critiquée et soumise à réserves, est la taxonomie numérique.
Cette approche quantitative de la taxonomie est définie par Peter H. A. Sneath et Robert Sokal comme "le groupement d'unités taxonomiques, en taxons à l'aide de méthodes numériques sur la base des états de leurs caractères".
La méthode consiste à réaliser un tableau de caractères, et à indiquer si chaque bactérie le possède ou non. Puis, à partir de ces données, on réalise une comparaison et des calculs de similarité. Prenons un exemple de calcul: le pourcentage de simple appariement:
Calcul des coefficients d'association de deux organismes:
Soit 4 ensembles a, b, c, d et deux bactéries isolées A et B:
* a: caractères présents chez A et B. (en rouge)
* b: caractères propres à la bactérie A. (bleu)
* c: caractères propres à la bactérie B. (vert)
* d: caractères manquant aux deux bactéries (beige)

Notion d'association de caractères
Simple appariement :
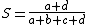
% de similarité :

Coefficient de Jaccard:

Les résultats sont exprimés sous forme de dendrogrammes: classification ascendante hiérarchique par niveau d'intégration.
Classification phylogénétique
En 1965, Pauling et Zuckerhandl suggèrent que les séquences d'acides nucléiques et protéiques changent avec le temps, et peuvent être considérées comme des chronomètres moléculaires.
Grâce à ce concept, il est donc possible de réaliser un arbre phylogénétique à partir de la comparaison de séquences définies chez différents organismes. En effet, on suppose qu'au cours du temps, ces séquences on été graduellement modifiées, si bien que des fonctions ont pu être perdue ou modifiées. Des séquences similaires se révélant très différentes chez deux groupes comparés montrent donc que ces deux groupes ont divergé voilà longtemps.
on choisira des séquences conservées chez tous les organismes pour réaliser une classification des grands groupes, et des séquences plus spécifiques pour réaliser une classification plus précise sur une famille, un genre, des populations ...
Pour les bactéries, nous savons que leurs ribosomes se composent de différents ARN ribosomiques, dont l'ARN 16S (S = Svedberg). Ni trop petit ni trop long, il a été choisi comme élément de comparaison au tout début de la taxonomie moléculaire. Il est digéré par la RNAse T1, et les fragments obtenus furent analysés en premier par Woese, obtenant l'arbre de Woese (voir figures ci-dessous):

ARN ribosomal 16S
A partir de ces comparaisons, il a été possible de dessiner un premier arbre phylogénique moléculaire. Woese réalisa cette construction:

Arbre phylogénétique universel
Etabli par comparaison de séquences d'ARNr
La position exacte des trois ensembles sur l'arbre est encore discutée, comme nous aurons occasion de le voir ultérieurement dans un article consacré au sujet, mais en attendant le lecteur intéressé par cet arbre peut se rapporter à l'article de G. J. Olsen & C.R. Woese: Ribosomal RNA: a key to Phylogeny. The FASEB Journal, 7, pp. 113-123, 1993.
Dans un arbre phylogénétique, on appelle noeud chaque point correspondant à un organisme ancêtre et branches les cheminements évolutifs ayant mené aux noeuds-fils. L'arbre peut avoir une échelle de temps ou de modifications moléculaires, alors représentées par la longueur des branches. Il peut être enraciné (montre un ancêtre commun) ou non (mettant en évidence uniquement les relations phylogénétiques).

Exemples d'arbres phylogénétiques enracinés (a) ou non (b)
Les résultats sont différents de la classification phénotypique ! Voici de nouveau l'exemple d'Escherichia coli:
* Règne: Procaryotes
* Embranchement / Division: Proteobacteria
* Classe: Gammaproteobacteriae
* Ordre: Enterobacteriales
* Famille: Enterobacteriaceae
* Genre: Escherichia
* Escherichia coli
Ces données ont été obtenues à partir de Pubmed Taxonomy browser.
Nous avons vu plus haut des exemples de protocoles expérimentaux et d'interprétation des résultats concernant la similitude entre deux bactéries A et B. Les caractères génétiques ainsi isolés peuvent également être retenus dans des matrices et donner des dendrogrammes !
La taxonomie polyphasique
De nombreux taxonomistes pensent que seule une combinaison des données génotypiques et phénotypiques peut permettre d'établir une phylogénie. C'est l'approche polyphasique.
Quand celà est possible, les taxonomistes s'emploient à utiliser un maximum d'informations, que ce soit des caractères écologiques, morphologiques, métaboliques comme des propriétés moléculaires, afin d'obtenir les résultats les plus fiables et les plus réalistes.
Sources:
* Prescott, Harley & Klein. Microbiologie. Editions DeBoeck (2003)
* Cours de microbiologie. Licence de Biochimie de Rennes 1 (2004)
* Pubmed taxonomy Browser (2006) [ En ligne ]

biologiste- Moderateur

- Messages : 140
inscrit(e) le: : 29/04/2009
Age : 35
Localisation : Sur terre
 Sujets similaires
Sujets similaires» Transgenèse bactérienne
» PHYSIOLOGIE & CROISSANCE [Bactérienne]
» Cours de Taxonomie animale
» Programme de Taxonomie animale pour les 2ème année Biologie cycle long
» PHYSIOLOGIE & CROISSANCE [Bactérienne]
» Cours de Taxonomie animale
» Programme de Taxonomie animale pour les 2ème année Biologie cycle long
DOC DZ...La Communauté des Etudiants et Praticiens en Médecine :: FORUMS VOISINS ... :: FILIERES VOISINES
:: les biologistes :: Taxonomie
Page 1 sur 1
Permission de ce forum:
Vous ne pouvez pas répondre aux sujets dans ce forum











» Cahiers d'anatomie
» précis d'obstétrique par robert merger
» 120 questions en gynécologie-obstétrique 4eme edition
» Gynécologie Obstétrique Fertilité & Sénologie , 2020
» Transfusion clinique et biologique 2020
» Banque de vidéos - Examens complémentaires en Pneumologie
» la collection Facile (ELSEVIER / MASSON)
» Dermatologie Guide pratique De la clinique au traitement ( livre scanné et complet)
» Coloscopie virtuelle, imagerie médicale pratique
» TOUTE L'EMBRYOLOGIE DE LA FECONDATION JUSQU'A L'INFINI!!!
» Histologie fonctionnelle Manuel et Atlas de Wheater, Exclusivité Doc DZ
» Dermatologie infectieuse
» plus de 3300 question à réponses ouvertes courtes de sémiologie
» ARBRES DECISIONNELS EN PEDIATRIE
» Imagerie des Urgences 2019
» Guide du bon usage examens d'imagerie médicale
» Recommandations de bonne pratique et Perfectionnement en Pediatrie en Exclusivité pour Vous
» Besoin d’un livre
» 18 cours de Pharmacologie Spéciale
» collection de cours orthopedie pour externes internes et residents
» LIVRE"Pathologies vasculaires oculaires" rapport SFO 2008
» Collection Clinique et Semiologie:
» Précis d'obstétrique
» Précis d'obstétrique
» Mémento de pathologie 5e édition 2017 Eclusivité Doc DZ
» appareil urinaire
» Merci de signaler les liens morts ici !
» La biochimie en 1001 QCM PCEM
» Livre Echographie 4 eme édition " imagerie médicale formation "
» Collection Cas cliniques en imagerie ...5 Livres pour vous
» Les Infections Intra-Abdominales Aiguës
» 50 cas clinique pediatrie.
» guide des medicaments injectables/adulte
» Protocoles médicaux
» physio humaine
» Comment télécharger nos documents et fichiers
» Guide de prise en charge de la diarhée chez l'enfant
» logiciel vidal expert 2013 avec activation jusqu'à 2100
» La Collection Campus illustré et référence : 8 livres pour vous...